Fecha de publicación: 03 Enero 2022
Estado: Sitio web eliminado. Recuperado desde WayBack Machine
Uno de los aspectos más difíciles de determinar en la identificación de patrones de las vacunas de c0r0n@v|rus es el método o procedimiento, por el cual los objetos que se vienen observando (micro/nano-routers, micro/nano-rectenas…), se han podido conformar. En la literatura científica se han encontrado multitud de trabajos que apuntaban a diversas técnicas de producción, tales como la litografía de electrones, haz de iones enfocado FIB (Focused Ion Beam) e incluso plantillas de ADN sintético, con las que se definirían los circuitos QCA de los nanorouters. Sin embargo, no se terminaban de encontrar pruebas claras de autoensamblaje en las muestras de la vacuna. Sin embargo, las sospechas, más que fundadas de este proceso, se confirmaron con la observación del vídeo elaborado por Ricardo Delgado el 27 de diciembre de 2021, en el que se presenciaba el movimiento de miles de partículas en una muestra de la vacuna Pfizer. Estas partículas parecían unirse para conformar estructuras más complejas, definiendo patrones geométricos sencillos, véase extracto en el siguiente vídeo 1.
Vídeo 1. Autoensamblaje observado en una muestra de la vacuna Pfizer. Fuente original: https://www.twitch.tv/videos/1245191848?t=00h34m56s (Delgado, R. 2021)
En la literatura científica, este comportamiento o movimiento casi dirigido de las partículas, en el contexto de la construcción de objetos y dispositivos micro/nano electrónicos en una nano-red intracorporal de comunicaciones, tenía altas probabilidades de corresponder a un proceso de autoensamblaje basado en ADN, crecimiento epitaxial y origami. Esta deducción resultó en la localización del articulado científico que, con alta probabilidad, podría confirmar el autoensamblaje de objetos complejos, incluyendo circuitos, placas, routers, sensores y otros componentes y dispositivos micro/nano electrónicos. Este descubrimiento explica cómo se auto-ensamblarían los componentes responsables del fenómeno de emisión de direcciones MAC bluetooth (Sarlangue, G.; Devilleger, J.; Trillaud, P.; Fouchet, S.; Taillasson, L.; Catteau, G. 2021). También explicaría el ensamblaje de nano-dispositivos, nano-sensores, nano-nodos, micro/nano-interfaces, micro/nano-routers, micro/nano-antenas, micro/nano-rectenas, con los que se configura el hardware de la red intra-corporal de nano-comunicaciones.
En la figura 1 se presentan los indicios de autoensamblaje observados en la literatura científica y su correspondencia con las muestras analizadas de la vacuna Pfizer. Desde un punto de vista morfológico, existen importantes coincidencias que permiten inferir y casi asumir que el autoensamblaje es una realidad constatable.
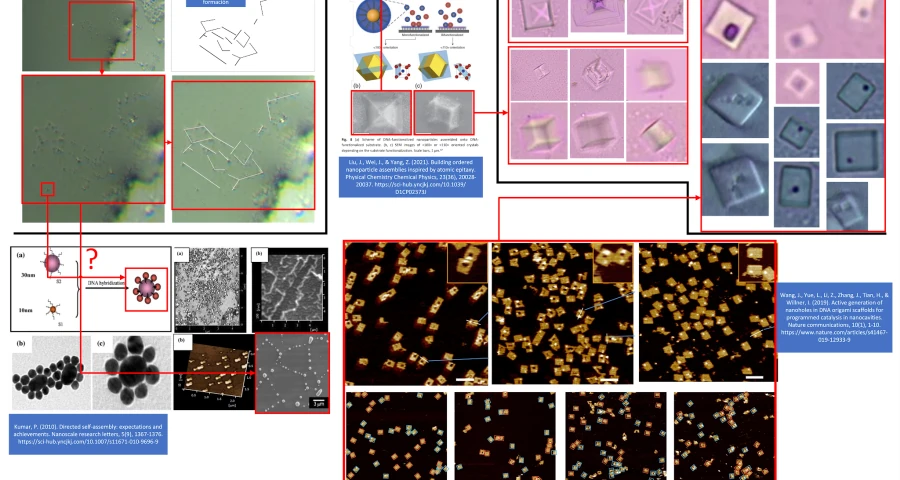
Debido a la complejidad de la temática de autoensamblaje, así como la relevancia de las evidencias descubiertas, se procederá a su análisis pormenorizado, en torno a tres epígrafes fundamentales: a) autoensamblaje dirigido; b) autoensamblaje mediante crecimiento epitaxial suave; c) autoensamblaje origami.
Autoensamblaje dirigido
El artículo de (Kumar, P. 2010) presenta el primer indicio claro de “autoensamblaje dirigido” que puede ser observado en la muestra de la vacuna, véase figura 2 y vídeo 1. Las nanopartículas que se observan, parecen unirse en clústeres de mayor tamaño y con ello, estructuras más complejas que se mueven en la gota de la muestra.

Según (Kumar, P. 2010), el autoensamblaje dirigido es clave para el desarrollo de dispositivos electrónicos, magnéticos y ópticos miniaturizados, lo cual encaja con los materiales derivados del grafeno hallados en las muestras de la vacuna, de hecho se afirma que “las nanopartículas han atraído una gran atención como tales componentes debido a sus propiedades únicas dependientes del tamaño, que incluyen super-paramagnetismo, quimioluminiscencia y catálisis. Para aprovechar al máximo las capacidades potenciales de las nanopartículas, necesitamos desarrollar nuevos métodos para ensamblarlas en patrones o estructuras útiles. Estas estructuras autoensambladas prometen nuevas oportunidades para desarrollar dispositivos ópticos, electrónicos, optoelectrónicos y magnéticos miniaturizados“.
Por otra parte, Kumar desvela que el método de “autoensamblaje dirigido” es el adecuado para poder generar dispositivos a escala nano y micro debido a su capacidad para usar puntos cuánticos o nanopuntos. Lo explica de la siguiente forma “a medida que el tamaño o las funciones del dispositivo se vuelven cada vez más pequeños, los procesos litográficos convencionales resultan ser limitados para su producción. Es necesario desarrollar métodos alternativos para sortear esta dificultad. A medida que se desarrollan tecnologías de fabricación convencionales, como la litografía óptica, también comienzan a tropezar con límites fundamentales… Además, se requieren nuevas técnicas de fabricación para ayudar a extender tanto la vida útil como el rango de aplicación de las técnicas existentes… La técnica de autoensamblaje dirigida se puede emplear de manera apropiada para producir nanoestructuras funcionales, por ejemplo, nanocables y una matriz organizada de nano-puntos (entiéndase puntos cuánticos)“. Dicho de otra forma, el “autoensamblaje dirigido” permite que los puntos cuánticos de un determinado material (por ejemplo el grafeno GQD Graphene Quantum Dots), se autoensamblen conforme a un patrón predefinido.
Entre los posibles tipos de autoensamblaje dirigido o guiado, Kumar reconoce el “ensamblaje guiado por plantillas donde utilizan patrones atómicos de superficie; el ensamblaje guiado por campo electromagnético o campo eléctrico, por haz de electrones, luz y láser, entre otros“. Además, reconoce que “el autoensamblaje dirigido es una técnica reproducible y robusta con perspectivas futuras para su uso a escala industrial… lo que significa construir estructuras bien ordenadas, a menudo intrigantes, lo que ha recibido mucha atención por su facilidad para organizar materiales en la nanoescala en estructuras ordenadas y producir estructuras complejas a gran escala“. Esto parece ser fundamental en el contexto de la red intra-corporal de nanocomunicaciones y nano/micro dispositivos, pues deben ser creados miles de dispositivos para su funcionamiento (Zhang, R.; Yang, K.; Abbasi, Q.H.; Qaraqe, K.A.; Alomainy, A. 2017 | Galal, A.; Hesselbach, X. 2018 | Galal, A.; Hesselbach, X. 2020).
Entre todas las formas de autoensamblaje, la más probable y que mayores coincidencias presenta a nivel morfológico es el autoensamblaje guiado por plantillas biológicas de ADN. Entre sus ventajas, Kumar destaca “la fabricación de nanocables ya que resuelven problemas de integración (eliminando la necesidad de manipular nanocables individuales). También se resuelven los problemas relacionados con los contactos para el transporte eléctrico y magnético“. Esto encaja con el tipo de nanodispositivos observados, por ejemplo las micro/nano rectenas y los materiales derivados del grafeno, los puntos cuánticos de grafeno GQD. De hecho, Kumar afirma que “el uso de plantillas físicas de ADN, da lugar al crecimiento de nanomateriales en una posición predefinida, eliminando la necesidad de manipulación posterior al crecimiento y proporcionando la facilidad de conexiones eléctricas para caracterizaciones adicionales“, lo cual ayuda a comprender cómo se construyen y definen las formas cuadrangulares observadas en las muestras de la vacuna, que guardan una gran semejanza con placas PCB, microchips, sensores y circuitos integrados. También añade que “tales plantillas dan lugar al crecimiento de nanopuntos (puntos cuánticos), nanocables verticales, que se pueden usar de manera controlable para fabricar dispositivos FET (Transistores de efecto campo), dispositivos de unión de túnel magnético y dispositivos para aplicaciones ópticas” lo cual viene a confirmar que con el autoensamblaje dirigido es posible crear nanotecnología miniaturizada de cualquier dispositivo electrónico conocido. Dicho de otra forma, el autoensamblaje guiado por plantillas biológicas de ADN, puede servir para confeccionar todos los dispositivos requeridos para una nano-red intracorporal, siendo factible que ésta sea la técnica empleada en las vacunas, a tenor de las imágenes observadas y las afirmaciones en la literatura científica (Catania, V.; Mineo, A.; Monteleone, S.; Patti, D. 2014 | Keren, K.; Berman, R.S.; Buchstab, E.; Sivan, U.; Braun, E. 2003).
Para más señas, Kumar indica que “las estrategias dirigidas por biomoléculas (plantillas biológicas de ADN) han demostrado ser muy prometedoras en el ensamblaje de nanopartículas en una amplia diversidad de arquitecturas, debido a su alta eficiencia, alta especificidad y programabilidad genética (McMillan, R.A.; Paavola, C.D.; Howard, J.; Chan, S.L.; Zaluzec, N.J.; Trent, J.D. 2002). Se ha demostrado que estos materiales nanoensamblados tienen aplicaciones potenciales en nuevos sistemas de detección, como biosensores (Taton, T.A.; Mirkin, C.A.; Letsinger, R.L. 2000) y sensores químicos (Liu, J.; Lu, Y. 2003 | Liu, J.; Lu, Y. 2006), y en la construcción de dispositivos nanoelectrónicos (Keren, K.; Berman, R.S.; Buchstab, E.; Sivan, U.; Braun, E. 2003) [configurados paradójicamente con nanotubos de carbono]” Lo que vuelve a confirmar que es una técnica/método conveniente en la implementación de nanotecnología en el cuerpo humano.
Autoensamblaje mediante crecimiento epitaxial suave
Si los indicios de autoensamblaje dirigido pueden considerarse, hipótesis perfectamente fundadas, el “autoensamblaje mediante crecimiento epitaxial suave” presenta pruebas aún más evidentes. En la figura 4 se observa una equivalencia exacta entre la literatura científica y las muestras de la vacuna Pfizer analizadas por el doctor (Campra, P. 2021). Algunos de los objetos más numerosos, con forma cuadrangular y piramidal, en realidad serían el resultado de una técnica de autoensamblaje epitaxial, que es de hecho, “uno de los procesos de fabricación de circuitos integrados” (Shen, J.; Sun, W.; Liu, D.; Schaus, T.; Yin, P. 2021 | Burns, M.A.; Mastrangelo, C.H.; Sammarco, T.S.; Man, F.P.; Webster, J.R.; Johnsons, B.N.; Burke, D.T. 1996 | Esener, S.C.; Hartmann, D.M.; Heller, M.J.; Cable, J.M. 1998 | Krahne, R.; Yacoby, A.; Shtrikman, H.; Bar-Joseph, I.; Dadosh, T.; Sperling, J. 2002 | Chen, Y.; Pepin, A. 2001). La epitaxia se refiere a la deposición de una capa de material (por ejemplo puntos cuánticos de grafeno, óxido de grafeno, hidrogel, etc.) sobre un sustrato primario de nucleación. Sin embargo, a diferencia de los procesos tradicionales de crecimiento, en este caso se consigue mediante la hibridación del ADN. Es en este punto, donde (Liu, J.; Wei, J.; Yang, Z. 2021) desarrolla uno de los objetos de su investigación.
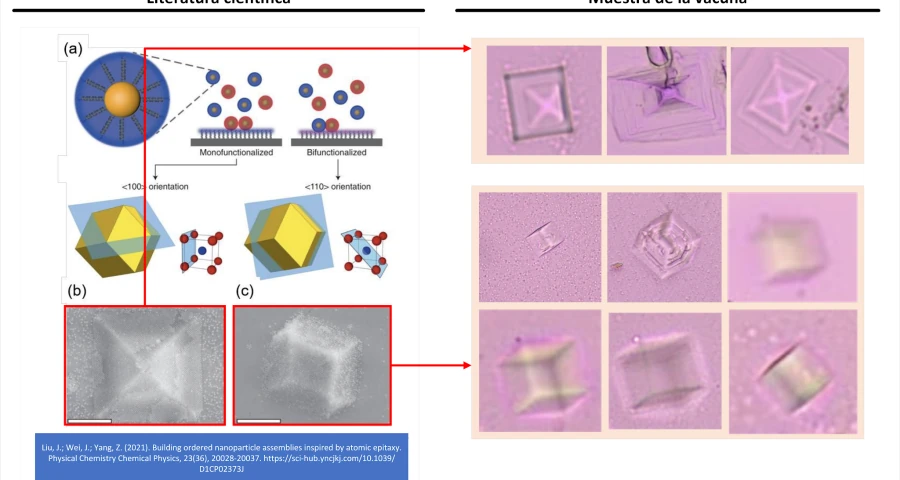
Según (Liu, J.; Wei, J.; Yang, Z. 2021) el autoensamblaje de “nanopartículas inorgánicas en ensamblajes de nanopartículas mesoscópicas o macroscópicas es una estrategia eficiente para fabricar dispositivos avanzados con funcionalidades emergentes a nanoescala. Además, el ensamblaje de nanopartículas en sustratos puede permitir la fabricación de dispositivos integrados en el sustrato, similar al crecimiento de cristales atómicos sobre un sustrato. El progreso reciente en el ensamblaje de nanopartículas sugiere que los ensamblajes de nanopartículas ordenados podrían producirse bien en un sustrato seleccionado, lo que se conoce como crecimiento epitaxial suave“. Esta definición viene a confirmar que la fabricación de micro/nano dispositivos electrónicos (circuitos integrados) puede llevarse a cabo mediante el crecimiento de cristales guiados en un sustrato o plantilla de ADN. Esto queda patente en la siguiente explicación “La hibridación de ADN se ha aplicado para ensamblar nanopartículas en superredes con estructuras cristalinas que son sorprendentemente ricas. Se encontró que la estructura tridimensional de doble hélice del ADN (paso fijo, diámetro fijo) tiene más ventajas que otros materiales para guiar las nanopartículas hacia un ensamblaje ordenado tridimensional (Nykypanchuk, D.; Maye, M.M.; Van-Der-Lelie, D.; Gang, O. 2008). El reconocimiento específico entre pares de bases y la capacidad de controlar la longitud de la cadena de ADN y la secuencia de bases, lo convierten en un arma poderosa para el ensamblaje a nanoescala. La capacidad de programación del ADN lo convierte en un ligando orientado a la estructura extremadamente atractivo“. Esto confirma que el autoensamblaje mediante ADN, no sólo permite construir estructuras 2D, ya que se pueden generar estructuras 3D gracias a los enlaces de la doble hélice del ADN, lo que permite usarlo para configurar todo tipo de formas, entre ellas las cúbicas y prismáticas que se observan en la figura 4.
Entre las experiencias citadas por (Liu, J.; Wei, J.; Yang, Z. 2021) cabe destacar el siguiente párrafo sobre el autoensamblaje epitexial, en el que se desvela una amplia experiencia en la experimentación de construcciones cristalinas basada en ADN, con una tolerancia al error (desajuste) de tan solo un 1%.
“Según (Lewis, D.J.; Zornberg, L.Z.; Carter, D.J.; Macfarlane, R.J. 2020) y sus colaboradores utilizaron esta técnica y una combinación de nanopartículas funcionalizadas con ADN y un sustrato funcionalizado por hebras de ADN para diseñar un proceso de ensamblaje epitaxial. Encontraron que las formas Winterbottom monocristalinas de cristales de nanopartículas se forman controlando las energías interfaciales entre los cristales y el fluido, el sustrato y el cristal, y el sustrato y el fluido. Otros ejemplos muestran que las nanopartículas injertadas de ADN autoensambladas en películas coloidales bidimensionales se pueden aplicar como sustrato para el ensamblaje epitaxial suave. Por ejemplo, según (Wang, M.X.; Seo, S.E.; Gabrys, P.A.; Fleischman, D.; Lee, B.; Kim, Y.; Mirkin, C.A. 2017) utilizaron nanopartículas recubiertas de ADN como elementos de construcción más elásticos y maleables para adaptarse mejor al desajuste de la red. Estudios posteriores (Gabrys, P.A.; Seo, S.E.; Wang, M.X.; Oh, E.; Macfarlane, R.J.; Mirkin, C.A. 2018) mostraron que las películas delgadas de superredes ensambladas por nanopartículas funcionalizadas con ADN, pueden almacenar cepas elásticas al deformarse y reorganizarse, con desajustes de red de hasta ± 7,7%, superando significativamente los desajustes de red de ± 1% permitidos por películas delgadas atómicas. Es importante destacar que estas nanopartículas recubiertas de ADN experimentan una relajación progresiva y coherente, disipando la tensión de forma elástica e irrecuperable mediante la formación de dislocaciones o vacantes. Por lo tanto, es posible cultivar películas coloidales heteroepitaxiales controlando equivalentes atómicos programables -blandos- de nanómetros y microestructuras utilizando nanocristales rígidos recubiertos con materiales poliméricos comprimibles blandos.” (Liu, J.; Wei, J.; Yang, Z. 2021)
Autoensamblaje origami
Finalmente, entre las formas de autoensamblaje más originales se encuentra el “método origami“, también vinculado al uso de plantillas de ADN. En este caso, los indicios se encuentran en el trabajo de (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019) titulado “Generación activa de nano-agujeros en andamios de origami de ADN para catálisis programada en nanocavidades“. El patrón de un punto o agujero dentro de una estructura cuadrangular, resulta llamativo y caracterizador desde el punto de vista morfológico. Este detalle se encontró en las imágenes obtenidas por el doctor Campra, lo que unido al objeto de estudio de autoensamblaje, permitió inferir que se trataba de otra pieza del puzle y que en realidad, deben existir objetos de mayor tamaño autoensamblados con el método origami. Las semejanzas son claras y evidentes, véase figura 5, ya que coincide la estructura cuadrangular de los objetos, la posición de los nano-agujeros inscritos dentro de la superficie, así como el número o cantidad de ellos observados en las muestras de la vacuna Pfizer.
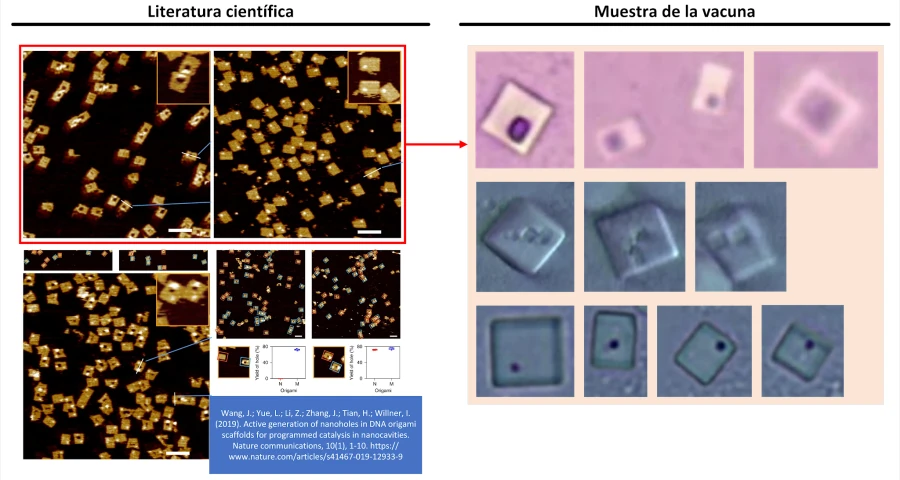
Pero, antes de proceder a analizar el asunto de los agujeros en los objetos cuadrangulares, merece la pena realizar una revisión de la introducción y estado de la cuestión que aportan los autores en su trabajo, ya que ayuda a situar las capacidades de la técnica y demostrar su vinculación a la nanotecnología empleada en las vacunas. De hecho se observan afirmaciones sorprendentes, ya que el autoensamblaje de origami es un “ensamblaje programado de nanoestructuras de ADN bidimensionales (2D) y tridimensionales (3D), que representan un avance importante en la nanotecnología del ADN” (Hong, F.; Zhang, F.; Liu, Y.; Yan, H. 2017 | Rothemund, P. W. 2006 | Endo, M.; Sugiyama, H. 2014), lo que confirma no sólo las posibles dimensiones o ejes de autoensamblaje, si no también que el método origami es compatible con el autoensamblaje de crecimiento epitexial suave y por consiguiente con el autoensamblaje dirigido o guiado. En todos los casos, el uso de las estructuras de ADN sintético, convenientemente configuradas, son los precursores necesarios para el desarrollo de las estructuras y objetos observados en las muestras de la vacuna.
Además, (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019) confirman que el método de autoensamblaje origami mediante ADN, permite el anclaje de componentes para configurar, entre otros dispositivos, antenas plasmónicas, identificadas anteriormente en las muestras de la vacuna como parte de la nano-red centrada en el cuerpo humano. Esto se afirma en la siguiente cita literal: “además de crear formas ingeniosas de estructuras de origami generadas por el plegamiento programado del ADN, las estructuras de origami se funcionalizaron con cadenas de ácido nucleico que sobresalen, o hebras de oligonucleótidos con bordes modificados. Las hebras que sobresalen se utilizaron como sitios de anclaje para la organización de los polímeros, proteínas y nanopartículas en los andamios de cada origami. Se demostraron funciones únicas de las nanoestructuras ensambladas en los andamios de origami, como el funcionamiento de las cascadas de enzimas, el diseño de antenas plasmónicas y el ensamblaje de estructuras quiroplasmónicas“. Esta explicación es fundamental para comprender el proceso de formación de superestructuras, guiadas por patrones de ADN, ya que se enlazan a través de las hebras que sobresalen de las piezas de construcción, funcionalizadas con nanopartículas (por ejemplo puntos cuánticos de grafeno), lo que unido al factor de escala y superconductor del material, proporcionan características plasmónicas, y de hall cuántico, lo que implica el autoensamblaje de transistores, y micro/nano chips de la complejidad que fuera precisa.
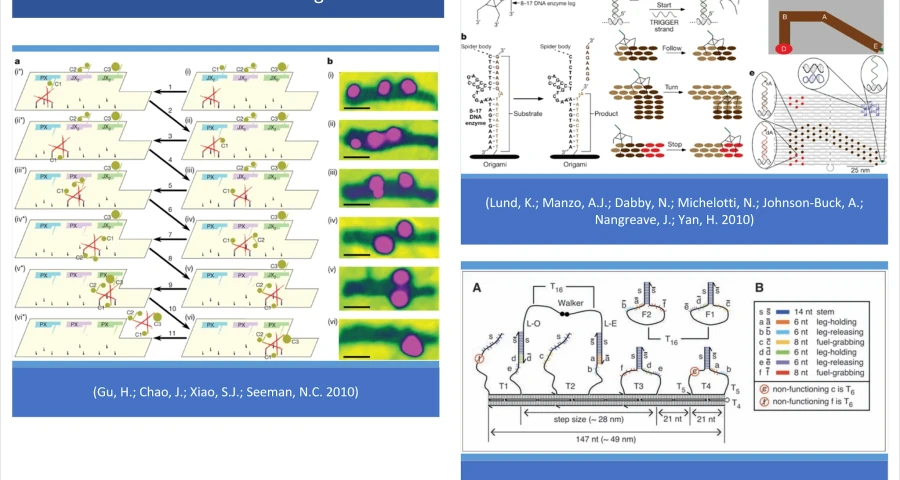
En su introducción, (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019) también aportan interesantes anotaciones y citas sobre las posibilidades de la técnica origami y el diseño de caminadores de ADN con capacidad motriz para iniciar movimiento, girar y detenerse, conforme a patrones de interacción moleculares. De hecho, según (Lund, K.; Manzo, A.J.; Dabby, N.; Michelotti, N.; Johnson-Buck, A.; Nangreave, J.; Yan, H. 2010) estos caminadores de ADN, son en esencia robots moleculares guiados por moléculas de sustrato (precursores) en un conjunto de estructuras (plantillas) de ADN origami. Esto queda confirmado en la siguiente cita a texto completo de Lund, corroborada también por (Omabegho, T.; Sha, R.; Seeman, N.C. 2009 | Gu, H.; Chao, J.; Xiao, S.J.; Seeman, N.C. 2010):
“Trasladar la robótica al nivel de una sola molécula es posible a priori, pero requiere enfrentar la capacidad limitada de las moléculas individuales para almacenar información y programas complejos. Una estrategia para superar este problema es utilizar sistemas que puedan obtener comportamientos complejos a partir de la interacción de robots simples con su entorno. Un primer paso en esta dirección fue el desarrollo de caminadores de ADN (DNA Walkers), que han pasado de ser no autónomos, a ser capaz de realizar movimientos dirigidos pero breves en pistas unidimensionales. En este trabajo demostramos que los caminantes aleatorios, también denominados arañas moleculares que comprenden una molécula de estreptavidina como un -cuerpo- inerte y tres desoxirribozimas como -patas- catalíticas, muestran un comportamiento robótico elemental cuando interactúan con un entorno definido con precisión. Las observaciones de microscopía de una sola molécula confirman que estos caminantes logran un movimiento direccional al detectar y modificar las pistas de las moléculas de sustrato dispuestas en un paisaje de origami de ADN bidimensional” (Lund, K.; Manzo, A.J.; Dabby, N.; Michelotti, N.; Johnson-Buck, A.; Nangreave, J.; Yan, H. 2010).
Esto puede confirmar la presencia de moléculas y piezas con capacidad para el autoensamblaje, su movimiento, orientación y auto-organización, para configurar dispositivos electrónicos complejos, conforme a los patrones y plantillas de ADN sintético, que se encuentran más próximas en una solución como la de la vacuna, tal como sugiere la observación del vídeo 1.
Continuando con el análisis de (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019), se agrega que “La funcionalización de bordes de los mosaicos de origami (de las plantillas de ADN), se aplicó para diseñar estructuras de origami multicomponente programadas y, en particular, para desarrollar dímeros de origami intercambiables“. Dicho de otra forma, las plantillas de ADN se pueden definir de forma tal, que se conformen con piezas concretas (partículas, proteínas, puntos cuánticos, etc.) de acuerdo a un programa o patrón predeterminado.
Sin embargo, la tecnología de origami de ADN puede abarcar otros ámbitos, tal como recoge las experiencias del estado de la cuestión de Wang y su equipo “se fabricaron ingeniosos sistemas de origami 3D. Por ejemplo, se ha demostrado el autoensamblaje de una caja de origami, el ensamblaje escalonado de estructuras de ADN programables a escala de gigadalton y el movimiento impulsado por la luz de paquetes de origami 3D para producir funciones quirópticas reversibles. Se sugirieron diferentes aplicaciones de las nanoestructuras de origami, incluida la catálisis programada, liberación controlada de fármacos, operaciones de puerta lógica y detección“. Entre las aplicaciones mencionadas, cabe destacar las operaciones de puerta lógica y detección, propias del diseño de circuitos QCA (Quantum Cell Automata) ya comentadas en la identificación de nanorouters entre los patrones observados en las vacunas. Esto es una prueba más de que la metodología origami de ADN es válida para desarrollar dispositivos electrónicos basados en puntos cuánticos, dada la capacidad para controlar la construcción ordenada de los cables y los circuitos.
Completada la revisión de los preámbulos del artículo de (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019), el discurso científico se centra en el objeto de las cavidades o agujeros en las “balsas de origami“, que en la vacuna se muestran como estructuras cuadrangulares con un punto inscrito dentro de su área. Según se indica “La mayoría de estas estructuras funcionales de origami implicaron, la modificación de abajo hacia arriba de las balsas de origami, la modificación del borde de las baldosas de origami o el plegado de las baldosas en tubos. Sin embargo, se puede considerar la funcionalización de estructuras de origami con nanocavidades (agujeros o barriles) que podrían actuar como contención o canales para transformaciones químicas guiadas. Hasta la fecha, tales cavidades se han fabricado dentro del ensamblaje pasivo de las baldosas de origami y estas cavidades se usaron para el acoplamiento específico del sitio de anticuerpos, la reconstitución de proteínas de membrana y la funcionalización de poros de estado sólido para transporte selectivo. Además, se han introducido estructuras de ADN (no origami) en las membranas y estas actuaron como canales para el transporte potencial estimulado de especies de cargas a través de las membranas. Por el contrario, el presente estudio introduce el concepto de fabricación activa de nanoagujeros en baldosas de origami. Informamos sobre la formación activa guiada por ADNzima de nanoagujeros en los andamios de origami y el desbloqueo mecánico molecular de los nanoagujeros levantando los dominios de ventana cubiertos. Al aplicar dos ADNzimas diferentes, se demuestra la fabricación programada y activada de nanoagujeros en las estructuras de origami. Además, utilizamos las cavidades en los diferentes andamios de origami como nanoambientes confinados para catálisis selectiva y específica. Además, destacamos un diseño para la apertura y cierre mecánico reversible por luz de los nanoagujeros, y la catálisis conmutable en las nanocavidades“. En esta explicación, que no deja duda de la intencionalidad de la manufactura de la técnica de origami, existe un detalle fundamental que debe ser considerado seriamente. Se trata de la capacidad de las cavidades en las estructuras de origami de ADN, para atrapar, inmovilizar y acoplar anticuerpos (Ouyang, X.; De-Stefano, M.; Krissanaprasit, A.; Bank-Kodal, A.L.; Bech-Rosen, C.; Liu, T.; Gothelf, K.V. 2017), que originalmente tendría la intencionalidad de servir para realizar estudios serológicos, pero que aplicado a la construcción de dispositivos electrónicos de escala micro/nano intracorporales, podría conseguir el objetivo de evitar la fagocitación e inmovilización de las estructuras autoconformadas. También se desvela que estos agujeros tienen un papel muy importante en la interacción con otras secuencias de ADN origami, que pueden encajar (como si de una pieza de Lego se tratase) para agregar nuevos andamios de construcción, tal como explican (Kurokawa, T.; Kiyonaka, S.; Nakata, E.; Endo, M.; Koyama, S.; Mori, E.; Mori, Y. 2018) en la figura 7.
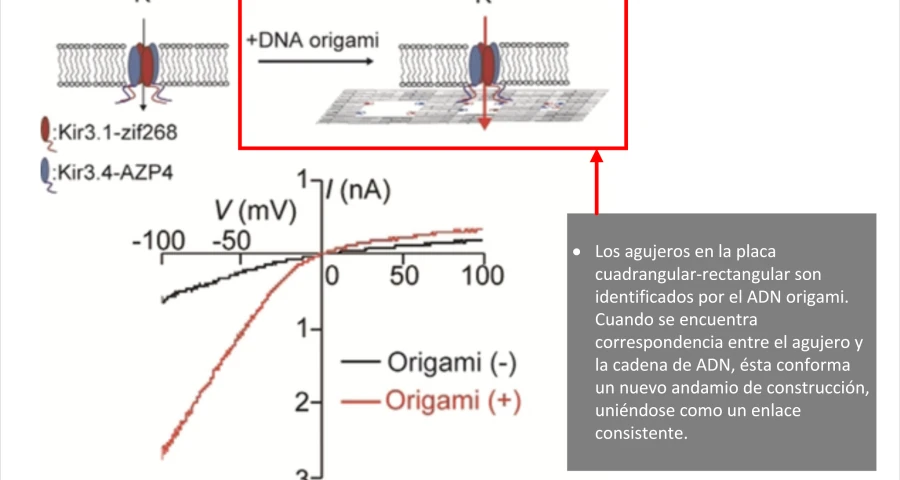
Otra de las aplicaciones citadas por Wang y su equipo para los agujeros es la de servir como canales o poros que atraviesan la placa o estructura de ADN origami, con la finalidad de desarrollar biosensores, tal como corroboran (Seifert, A.; Göpfrich, K.; Burns, J.R.; Fertig, N.; Keyser, U.F.; Howorka, S. 2015 | Burns, J.R.; Seifert, A.; Fertig, N.; Howorka, S. 2016). De hecho se indica que “Los nanoporos que atraviesan la membrana a partir de ADN plegado son un ejemplo reciente de nanoestructuras biomiméticas artificiales que pueden abrir aplicaciones en biosensores, administración de fármacos y nanofluidos… Establecemos que los poros del ADN exhiben dos estados de conductancia dependientes del voltaje. Los voltajes transmembrana bajos favorecen un nivel estable de alta conductancia, que corresponde a un poro de ADN sin obstrucciones. La anchura interior esperada del canal abierto se confirma midiendo el cambio de conductancia en función del tamaño del poli (etilenglicol) (PEG), por lo que se supone que entran PEG más pequeños en el poro“. Esto no sólo encaja con uno de los componentes declarados en la lista de excipientes de la vacuna Pfizer, ya que también concuerda con la conductividad necesaria para los componentes de la nano-red orientada al cuerpo humano (Yang, J.; Ma, M.; Li, L.; Zhang, Y.; Huang, W.; Dong, X. 2014 | Abbasi, Q. H.; Yang, K.; Chopra, N.; Jornet, J.M.; Abuali, N.A.; Qaraqe, K.A.; Alomainy, A. 2016 | Oukhatar, A.; Bakhouya, M.; El Ouadghiri, D. 2021)
Bibliografía
- Abbasi, Q. H.; Yang, K.; Chopra, N.; Jornet, J.M.; Abuali, N.A.; Qaraqe, K.A.; Alomainy, A. (2016). Nano-communication for biomedical applications: A review on the state-of-the-art from physical layers to novel networking concepts. IEEE Access, 4, pp. 3920-3935. https://doi.org/10.1109/ACCESS.2016.2593582
- Burns, J.R.; Seifert, A.; Fertig, N.; Howorka, S. (2016). Un canal biomimético basado en ADN para el transporte controlado por ligando de carga molecular cargada a través de una membrana biológica = A biomimetic DNA-based channel for the ligand-controlled transport of charged molecular cargo across a biological membrane. Nature nanotechnology, 11(2), pp. 152-156. https://doi.org/10.1038/nnano.2015.279
- Burns, M.A.; Mastrangelo, C.H.; Sammarco, T.S.; Man, F.P.; Webster, J.R.; Johnsons, B.N.; Burke, D.T. (1996). Estructuras microfabricadas para análisis de ADN integrado = Microfabricated structures for integrated DNA analysis. Proceedings of the National Academy of Sciences, 93(11), pp. 5556-5561. https://doi.org/10.1073/pnas.93.11.5556
- Campra, P. (2021a). Observaciones de posible microbiótica en vacunas COVID RNAm Version 1. https://corona2inspect.net/documentacion-y-publicaciones-del-doctor-pablo-campra-madrid/campra-p-2021-oct-microscopic-objects-frequently-observed-in-mrna-covid-19-vaccines/ | http://dx.doi.org/10.13140/RG.2.2.13875.55840
- Campra, P. (2021b). Detección de grafeno en vacunas COVID19 por espectroscopía Micro-RAMAN. https://corona2inspect.net/documentacion-y-publicaciones-del-doctor-pablo-campra-madrid/campra-p-2021-nov-deteccion-de-grafeno-en-vacunas-covid-19-por-espectroscopia-micro-raman/
- Campra, P. (2021c). MICROSTRUCTURES IN COVID VACCINES: ¿inorganic crystals or Wireless Nanosensors Network? https://corona2inspect.net/documentacion-y-publicaciones-del-doctor-pablo-campra-madrid/campra-p-2021-nov-microstructures-in-covid-vaccines-inorganic-crystals-or-wireless-nanosensors-network/
- Catania, V.; Mineo, A.; Monteleone, S.; Patti, D. (2014). Descubrimiento de topología distribuida en nano redes en chip autoensambladas = Distributed topology discovery in self-assembled nano network-on-chip. Computers & Electrical Engineering, 40(8), pp. 292-306. https://doi.org/10.1016/j.compeleceng.2014.09.003
- Chen, Y.; Pepin, A. (2001). Nanofabricación: métodos convencionales y no convencionales = Nanofabrication: Conventional and nonconventional methods. Electrophoresis, 22(2), pp. 187-207. https://doi.org/10.1002/1522-2683(200101)22:2%3C187::AID-ELPS187%3E3.0.CO;2-0
- Delgado, R. (2021). Directo del contenido de una vacuna Pfizer al microscopio [Programa especial]. La Quinta Columna. https://www.twitch.tv/videos/1245191848
- Endo, M.; Sugiyama, H. (2014). Imágenes de una sola molécula de movimientos dinámicos de biomoléculas en nanoestructuras de origami de ADN utilizando microscopía de fuerza atómica de alta velocidad = Single-molecule imaging of dynamic motions of biomolecules in DNA origami nanostructures using high-speed atomic force microscopy. Accounts of chemical research, 47(6), pp. 1645-1653. https://doi.org/10.1021/ar400299m
- Esener, S.C.; Hartmann, D.M.; Heller, M.J.; Cable, J.M. (1998). Microensamblaje asistido por ADN: una tecnología de integración heterogénea para optoelectrónica = DNA-assisted microassembly: a hetrogeneous integration technology for optoelectronics. En: Heterogeneous Integration: Systems on a Chip: A Critical Review (Vol. 10292, p. 1029208). International Society for Optics and Photonics. https://doi.org/10.1117/12.300616
- Gabrys, P.A.; Seo, S.E.; Wang, M.X.; Oh, E.; Macfarlane, R.J.; Mirkin, C.A. (2018). Discrepancia de celosía en películas delgadas de nanopartículas cristalinas = Lattice mismatch in crystalline nanoparticle thin films. Nano letters, 18(1), pp. 579-585. https://doi.org/10.1021/acs.nanolett.7b04737
- Galal, A.; Hesselbach, X. (2018). Arquitectura de comunicación de nano-redes: modelado y funciones = Nano-networks communication architecture: Modeling and functions. Nano Communication Networks, 17, pp. 45-62. https://doi.org/10.1016/j.nancom.2018.07.001
- Galal, A.; Hesselbach, X. (2020). Protocolo de descubrimiento de ruta basado en probabilidades para nano-redes electromagnéticas = Probability-based path discovery protocol for electromagnetic nano-networks. Computer Networks, 174, 107246. https://doi.org/10.1016/j.comnet.2020.107246
- Gu, H.; Chao, J.; Xiao, S.J.; Seeman, N.C. (2010). Una línea de montaje de nanoescala de ADN programable basada en proximidad = A proximity-based programmable DNA nanoscale assembly line. Nature, 465(7295), pp. 202-205. https://doi.org/10.1038/nature09026
- Hong, F.; Zhang, F.; Liu, Y.; Yan, H. (2017). Origami de ADN: andamios para crear estructuras de orden superior = DNA origami: scaffolds for creating higher order structures. Chemical reviews, 117(20), pp. 12584-12640. https://doi.org/10.1021/acs.chemrev.6b00825
- Keren, K.; Berman, R.S.; Buchstab, E.; Sivan, U.; Braun, E. (2003). DNA-Templated Carbon Nanotube Field-Effect Transistor = Transistor de efecto de campo de nanotubos de carbono con plantilla de ADN. Science , 302 (5649), pp. 1380-1382. https://doi.org/10.1126/science.1091022 | https://sci-hub.yncjkj.com/10.1126/science.1091022
- Krahne, R.; Yacoby, A.; Shtrikman, H.; Bar-Joseph, I.; Dadosh, T.; Sperling, J. (2002). Fabricación de huecos a nanoescala en circuitos integrados = Fabrication of nanoscale gaps in integrated circuits. Applied physics letters, 81(4), pp. 730-732. https://doi.org/10.1063/1.1495080
- Kumar, P. (2010). Autoensamblaje dirigido: expectativas y logros = Directed self-assembly: expectations and achievements. Nanoscale research letters, 5(9), pp. 1367-1376. https://doi.org/10.1007/s11671-010-9696-9 | https://sci-hub.yncjkj.com/10.1007/s11671-010-9696-9
- Kurokawa, T.; Kiyonaka, S.; Nakata, E.; Endo, M.; Koyama, S.; Mori, E.; Mori, Y. (2018). Andamios de ADN Origami como plantillas para canales funcionales tetraméricos Kir3 K+ = DNA origami scaffolds as templates for functional tetrameric Kir3 K+ channels. Angewandte Chemie International Edition, 57(10), pp. 2586-2591. https://doi.org/10.1002/anie.201709982
- Lewis, D.J.; Zornberg, L.Z.; Carter, D.J.; Macfarlane, R.J. (2020). Construcciones Winterbottom monocristalinas de superredes de nanopartículas = Single-crystal Winterbottom constructions of nanoparticle superlattices. Nature materials, 19(7), pp. 719-724. https://doi.org/10.1038/s41563-020-0643-6
- Liu, J.; Lu, Y. (2003). Un biosensor de plomo colorimétrico que utiliza un ensamblaje de nanopartículas de oro dirigido por ADNzima = A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. Journal of the American Chemical Society, 125(22), pp. 6642-6643. https://doi.org/10.1021/ja034775u
- Liu, J.; Lu, Y. (2006). Detección colorimétrica rápida de adenosina y cocaína basada en un diseño de sensor general que incluye aptámeros y nanopartículas = Fast colorimetric sensing of adenosine and cocaine based on a general sensor design involving aptamers and nanoparticles. Angewandte Chemie, 118(1), pp. 96-100. https://doi.org/10.1002/ange.200502589
- Liu, J.; Wei, J.; Yang, Z. (2021). Construyendo ensamblajes ordenados de nanopartículas inspirados en la epitaxia atómica = Building ordered nanoparticle assemblies inspired by atomic epitaxy. Physical Chemistry Chemical Physics, 23(36), pp. 20028-20037. https://doi.org/10.1039/D1CP02373J | https://sci-hub.yncjkj.com/10.1039/D1CP02373J
- Lund, K.; Manzo, A.J.; Dabby, N.; Michelotti, N.; Johnson-Buck, A.; Nangreave, J.; Yan, H. (2010). Robots moleculares guiados por paisajes prescriptivos = Molecular robots guided by prescriptive landscapes. Nature , 465 (7295), pp. 206-210. https://doi.org/10.1038/nature09012
- McMillan, R.A.; Paavola, C.D.; Howard, J.; Chan, S.L.; Zaluzec, N.J.; Trent, J.D. (2002). Matrices de nanopartículas ordenadas formadas en plantillas de proteína chaperonina diseñadas = Ordered nanoparticle arrays formed on engineered chaperonin protein templates. Nature materials, 1(4), pp. 247-252. https://doi.org/10.1038/nmat775
- Nykypanchuk, D.; Maye, M.M.; Van-Der-Lelie, D.; Gang, O. (2008). Cristalización guiada por ADN de nanopartículas coloidales = DNA-guided crystallization of colloidal nanoparticles. Nature, 451(7178), pp. 549-552. https://doi.org/10.1038/nature06560
- Omabegho, T.; Sha, R.; Seeman, N.C. (2009). Un motor browniano de ADN bípedo con patas coordinadas = A bipedal DNA Brownian motor with coordinated legs. Science, 324(5923), pp. 67-71. https://doi.org/10.1126/science.1170336
- Oukhatar, A.; Bakhouya, M.; El Ouadghiri, D. (2021). Red de nano sensores inalámbricos de base electromagnética: arquitecturas y aplicaciones = Electromagnetic-Based Wireless Nano-Sensors Network: Architectures and Applications. J. Commun., 16(1), 8. http://dx.doi.org/10.12720/jcm.16.1.8-19
- Ouyang, X.; De-Stefano, M.; Krissanaprasit, A.; Bank-Kodal, A.L.; Bech-Rosen, C.; Liu, T.; Gothelf, K.V. (2017). Acoplamiento de anticuerpos en las cavidades de las estructuras de origami de ADN = Docking of antibodies into the cavities of DNA origami structures. Angewandte Chemie, 129(46), pp. 14615-14619. https://doi.org/10.1002/ange.201706765
- Rothemund, P. W. (2006). ADN plegable para crear formas y patrones a nanoescala = Folding DNA to create nanoscale shapes and patterns. Nature, 440(7082), pp. 297-302. https://doi.org/10.1038/nature04586
- Sarlangue, G.; Devilleger, J.; Trillaud, P.; Fouchet, S.; Taillasson, L.; Catteau, G. (2021). Objetivación de la existencia de direcciones MAC detectables en la gama de frecuencias de Bluetooth tras una inoculación de la terapia con antígeno COVID y la prueba PCR de detección de COVID. https://gloria.tv/post/vHCyNRQ246TC3jehvWkAVt2GG
- Seifert, A.; Göpfrich, K.; Burns, J.R.; Fertig, N.; Keyser, U.F.; Howorka, S. (2015). Nanoporos de ADN bicapa con cambio de voltaje entre estado abierto y cerrado = Bilayer-spanning DNA nanopores with voltage-switching between open and closed state. ACS nano, 9(2), pp. 1117-1126. https://doi.org/10.1021/nn5039433
- Shen, J.; Sun, W.; Liu, D.; Schaus, T.; Yin, P. (2021). Nanolitografía tridimensional guiada por epitaxia modular de ADN = Three-dimensional nanolithography guided by DNA modular epitaxy. Nature Materials, 20(5), pp. 683-690. https://doi.org/10.1038/s41563-021-00930-7
- Taton, T.A.; Mirkin, C.A.; Letsinger, R.L. (2000). Detección escanométrica de matrices de ADN con sondas de nanopartículas = Scanometric DNA array detection with nanoparticle probes. Science, 289(5485), pp. 1757-1760. https://doi.org/10.1126/science.289.5485.1757
- Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. (2019). Generación activa de nanoagujeros en andamios de origami de ADN para catálisis programada en nanocavidades = Active generation of nanoholes in DNA origami scaffolds for programmed catalysis in nanocavities. Nature communications, 10(1), pp. 1-10. https://doi.org/10.1038/s41467-019-12933-9
- Wang, M.X.; Seo, S.E.; Gabrys, P.A.; Fleischman, D.; Lee, B.; Kim, Y.; Mirkin, C.A. (2017). Epitaxia: equivalentes átomos programables versus átomos = Epitaxy: Programmable atom equivalents versus atoms. ACS nano, 11(1), pp. 180-185. https://doi.org/10.1021/acsnano.6b06584
- Yang, J.; Ma, M.; Li, L.; Zhang, Y.; Huang, W.; Dong, X. (2014). Nanomallas de grafeno: nuevos materiales versátiles = Graphene nanomesh: new versatile materials. Nanoscale, 6(22), pp. 13301-13313. https://doi.org/10.1039/C4NR04584J | https://sci-hub.yncjkj.com/10.1039/c4nr04584j
- Zhang, R.; Yang, K.; Abbasi, Q.H.; Qaraqe, K.A.; Alomainy, A. (2017). Caracterización analítica de la nanored In-Vivo de Terahercios en presencia de interferencia basada en el esquema de comunicación TS-OOK = Analytical characterisation of the terahertz in-vivo nano-network in the presence of interference based on TS-OOK communication scheme. IEEE Access, 5, pp. 10172-10181. https://doi.org/10.1109/ACCESS.2017.2713459
